皮爾遜積差相關係數在此系列文的EDA文章內有出現過幾次了,他其實就是pandas dataframe的corr方法產生的結果:
#Pearson correlation
dataset.corr()
皮爾遜積差相關係數: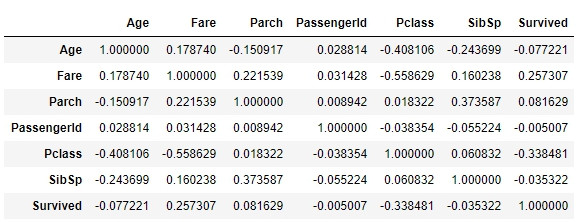
Pearson相關係數測量表徵之間的線性關係。係數的值在-1和+1之間變化,其中0表示它們之間沒有相關性。 接近-1或+1的相關性意味著極強的線性關係。pandas .corr()方法計算每一個表徵和其餘所有表徵的皮爾遜積差相關係數。由於人類是視覺化的動物,直接查看皮爾遜積差相關係數可能會難以消化,因此所以在過去的EDA文章當中,我總是使用熱度圖來視覺化皮爾遜積差相關係數:
熱度圖使用了顏色來做關聯性的區分;熱度圖中顏色越極端(接近全白、全黑)的格子代表關聯性越強的表徵。熱度圖中有一條對角的全白線,此現象應該忽略,因為它是代表表徵與他自己本身的關聯性自然為1。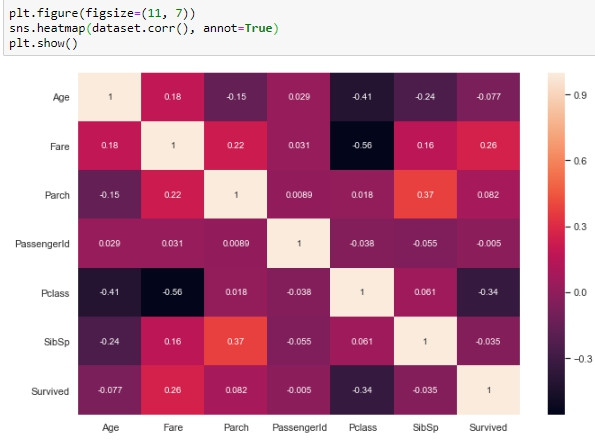
而以titanic的資料級為例,當我們只想要觀察生存率與其他表徵的關聯性,我們就可以省略顯示其他的表徵,只看該表中生存率與其他表徵的皮爾遜積差相關係數: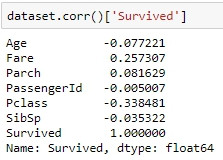
使用pandas的操作,我們能夠進一步的對係數做篩選,也就是對係數做閾值限定。這麼做的好處是有時候我們僅關心對目標表徵關聯性較強的其他表徵,而這也是我們此篇涵蓋對特徵選擇的重點;我們將以皮爾遜積差相關係數配合閾值來做特徵選擇。
以下示範只保留皮爾遜積差相關係數絕對值大於0.1的表徵名稱: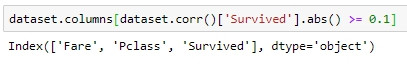
藉此,我們能夠列舉出自訂的皮爾遜積差相關係數閾值,驗證各種閾值下對模型的改善程度,得到表現最好的特徵組合:
thresholds = [0.04, 0.05, 0.06, 0.07, 0.08, 0.09, 0.1, 0.11, 0.12]
for threshold in thresholds:
highly_corr_features = dataset.columns[dataset.corr()['Survived'].abs() > threshold]
highly_corr_features = highly_corr_features.drop('Survived')
dummies_x_subsetted = dataset[highly_corr_features]
random_forest = RandomForestClassifier()
train_x, valid_x, train_y, valid_y = train_test_split(dataset, y, test_size=0.1)
random_forest.fit(train_x, train_y)
print(random_forest.score(valid_x, valid_y))
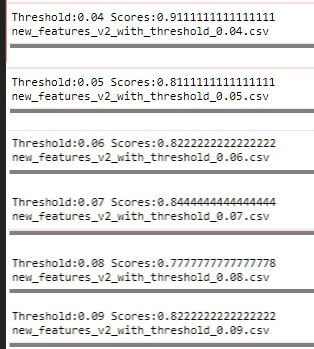
觀察結果得出的結論為,當閾值為0.04時模型的表現最好,這就是我們的特徵選擇依據。也看一下所有關聯係數在0.04以上的表徵,為Age/Fare/Parch/Pclass/Survived,但是注意要排除Survived。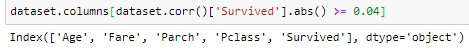
作為特徵選擇眾多方法中其中一種,皮爾遜積差相關係數的特徵選擇是一個很簡便但是有用的方法。下一篇文章將探討使用假設進行特徵選擇。
